CRYSOFT – IC BARI
DESCRIZIONE DELLE ATTIVITA’ DI RICERCA E RIFERIMENTI DELLA PERSONA DI CONTATTO:
Sviluppo di metodologie computazionali per la determinazione strutturale di molecole di interesse biologico o biotecnologico a diversa complessità mediante cristallografia (CrySoft)
Le metodologie cristallografiche sono di fondamentale importanza per lo studio della materia condensata. Esse consentono di dedurre la struttura cristallina partendo dai dati di diffrazione. Le proprietà fisiche, chimiche e biologiche dei vari composti dipendono fortemente dalla posizioni relative degli atomi all’interno delle molecole.
L’attività del gruppo di ricerca CrySoft si colloca a cavallo tra le aree tematiche di chimica e biologia strutturale ed è rivolta allo sviluppo di metodi cristallografici innovativi per la soluzione strutturale ab-initio
e non di composti di varia complessità, da dati di diffrazione (RX o elettroni) da cristallo singolo, alla implementazione degli algoritmi proposti in programmi di calcolo automatico e all’applicazione delle nuove procedure a dati sperimentali (ad es. determinazione strutturale di farmaci o materiali di interesse biologico e tecnologico). Tale attività entra a pieno titolo nella tradizione dell’Istituto di Cristallografia, leader scientifico a livello internazionale per le teorie sviluppate e per il relativo software.
Nell’ambito degli studi riguardanti le piccole/medie molecole (ad esempio farmaci o ligandi) l’attività si è concentrata sul continuo potenziamento degli algoritmi di fasazione ab initio nello spazio reciproco (Metodi Diretti) e sul miglioramento del processo di estensione e affinamento delle fasi nello spazio diretto, ottenuto modificando gli algoritmi coinvolti nella procedura di ‘direct space refinement’ (DSR).
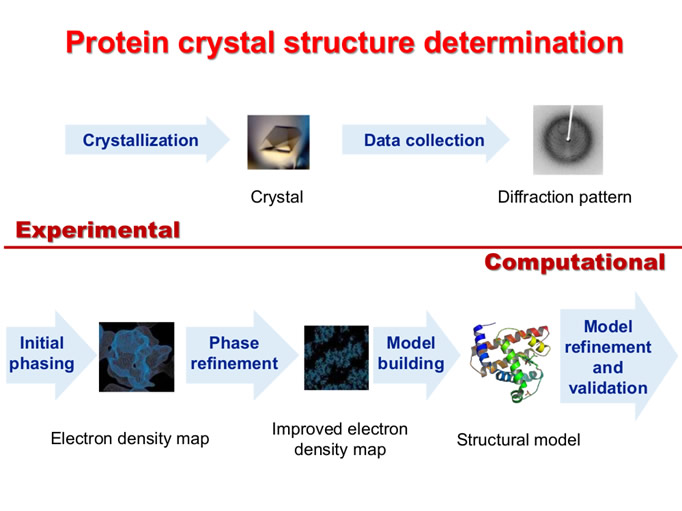
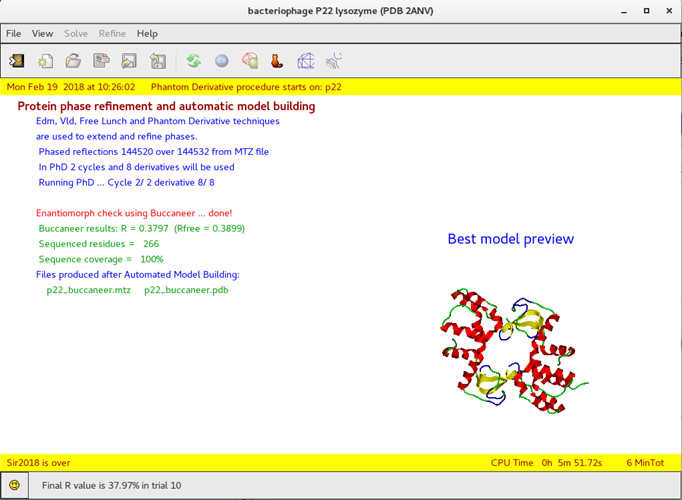
Per quanto riguarda la determinazione strutturale delle macromolecole, caratterizzate da dati spesso incompleti e a bassa risoluzione, essa è quasi sempre problematica e non ancora completamente automatizzata. Le ricerche in questo ambito vertono sullo studio di nuove procedure atte a favorire la risoluzione di proteine introducendo, nelle diverse tecniche utilizzate (ab initio, MAD/SAD, MIR/SIR, Molecular Replacement in sinergia con il metodo Phantom Derivative), le procedure innovative frutto degli sviluppi teorici elaborati dall’IC.
Tutto ciò ha portato ad aggiornare i diversi algoritmi di soluzione strutturale (con dati da cristallo singolo) implementati nei package automatici Sir2018 e Il Milione, per offrire alla comunità internazionale un software efficiente, flessibile, performante e di facile utilizzo.
I programmi prodotti, gratuiti per il mondo accademico, sono utilizzati proficuamente da migliaia di ricercatori in tutto il mondo.
Referente dell’Attività: GIOVANNI LUCA CASCARANO – gianluca.cascaranoATic.cnr.it
PERSONALE COINVOLTO:
Personale strutturato:
Benedetta Carrozzini, Rocco Caliandro, Giovanni Luca Cascarano
Associati:
Carmelo Giacovazzo, Maria Cristina Burla, Giampiero Polidori
FACILITIES E LABORATORI A DISPOSIZIONE:
(Contenuto non presente al momento)
PROGETTI DI RICERCA NEL GRUPPO:
(Contenuto non presente al momento)
PUBBLICAZIONI NEL GRUPPO:
- Burla M.C., Carrozzini B., Cascarano G.L., Giacovazzo C. & Polidori G. (2017) – Solving proteins at non-atomic resolution by Direct Methods: update – Jour. Appl. Cryst., 50, 1048-1055.
- Burla M.C., Carrozzini B., Cascarano G.L., Giacovazzo C. & Polidori G. (2015) – Solving protein at non-atomic resolution by Direct Methods- Jour. Appl. Cryst., 48, 1692-1698.
- Burla M.C., Carrozzini B., Cascarano G.L., Giacovazzo C. & Polidori G. (2015) – Refining a model electron-density map via the Phantom Derivative method – Acta Cryst., D71, 1864-1871.
- Burla M.C., Caliandro R., Carrozzini B., Cascarano G.L., Cuocci C., Giacovazzo C., Mallamo M., Mazzone A. & Polidori G. (2015) – Crystal structure determination and refinement via SIR2014- Jour. Appl. Cryst., 48, 306-309.
- Caliandro R., Carrozzini B., Cascarano G.L., Comunale G., Giacovazzo C. & Mazzone A. (2014) – Protein phasing at non-atomic resolution by combining Patterson and VLD techniques. – Acta Cryst., D70,