Sviluppo ed applicazione di metodologie cristallografiche per la determinazione strutturale di molecole di interesse biologico o tecnologico di varia complessità
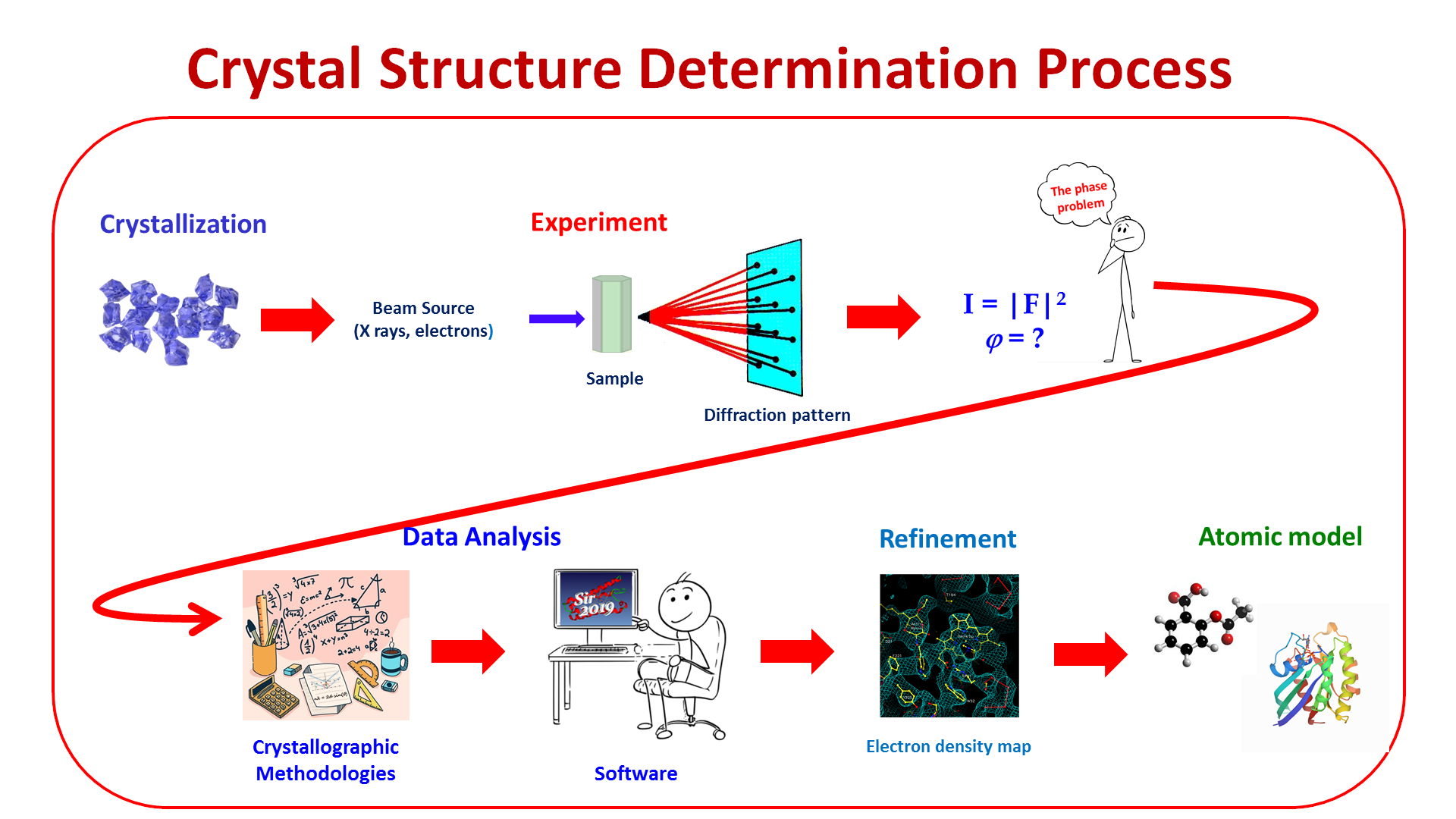
Sviluppo e applicazione di metodi computazionali per la soluzione strutturale di composti di varia natura e complessità, partendo da dati di diffrazione (raggi X o elettroni) da cristallo singolo, con particolare riguardo a molecole di interesse biologico o tecnologico.
Le metodologie cristallografiche sono fondamentali per lo studio delle proprietà fisiche e chimiche o delle funzionalità biologiche di composti di natura e complessità differenti (dalle piccole molecole alle proteine), poiché esse dipendono fortemente dalla disposizione degli atomi all’interno delle molecole.
La soluzione strutturale di piccole molecole può essere oggi considerata un processo di routine, sebbene non banale; al contrario la determinazione strutturale di proteine o acidi nucleici, i cui dati sono spesso incompleti e a bassa risoluzione, è ancora un processo complicato e non completamente automatizzato.
Questa attività di ricerca è rivolta:
– allo sviluppo di metodi cristallografici innovativi per la soluzione strutturale di materiali cristallini a diversa complessità, focalizzando l’attenzione sulla realizzazione di specifiche pipeline (dalla fasazione alla ricostruzione automatica del modello strutturale) dedicate alle macromolecole, basate su differenti approcci: Ab Initio, MAD/SAD e in particolare Molecular Replacement (il più utilizzato);
– ad un costante aggiornamento dei diversi algoritmi di soluzione strutturale implementati nel software SIR20XX (prodotto dai ricercatori dell’IC), per continuare ad offrire alla comunità internazionale un programma efficiente, flessibile, performante e di facile utilizzo, già oggi diffusamente e proficuamente utilizzato in migliaia di laboratori in tutto il mondo.
– all’applicazione delle nuove procedure a casi reali.
– Burla M.C., Carrozzini B., Cascarano G.L., Giacovazzo C. & Polidori G. (2020) – Cyclic automated model building (CAB) applied to nucleic acids – Crystals, 10, 280. Doi: 10.3390/cryst10040280
– Burla M.C., Carrozzini B., Cascarano G.L., Giacovazzo C. & Polidori G. (2020) – How far are we from automatic crystal structure solution via molecular-replacement techniques? – Acta. Cryst., D76, 9-18. Doi: 10.1107/S2059798319015468
– Burla M.C., Carrozzini B., Cascarano G.L., Giacovazzo C. & Polidori G. (2018) – CAB: a cyclic automatic model-building procedure – Acta. Cryst., D74, 1096-1104. Doi: 10.1107/S2059798318013438
– Burla M.C., Caliandro R., Carrozzini B., Cascarano G.L., Cuocci C., Giacovazzo C., Mallamo M., Mazzone A. & Polidori G. (2015) – Crystal structure determination and refinement via SIR2014- Jour. Appl. Cryst., 48, 306-309. Doi: 10.1107/S1600576715001132


